宏基因组学通常涉及对只能被描述为“棘手”的 DNA 样本进行测序。这种 DNA 显示出高度的异质性,这会导致种间错误组装。这些错误组装威胁到宏基因组测序的目的,即通过在给定样本中生成多个基因组草图来全面研究基因库。由于这些样本中存在某些无法使用标准微生物学技术进行培养的生物,这个问题变得更加复杂。如何解决这一系列问题?
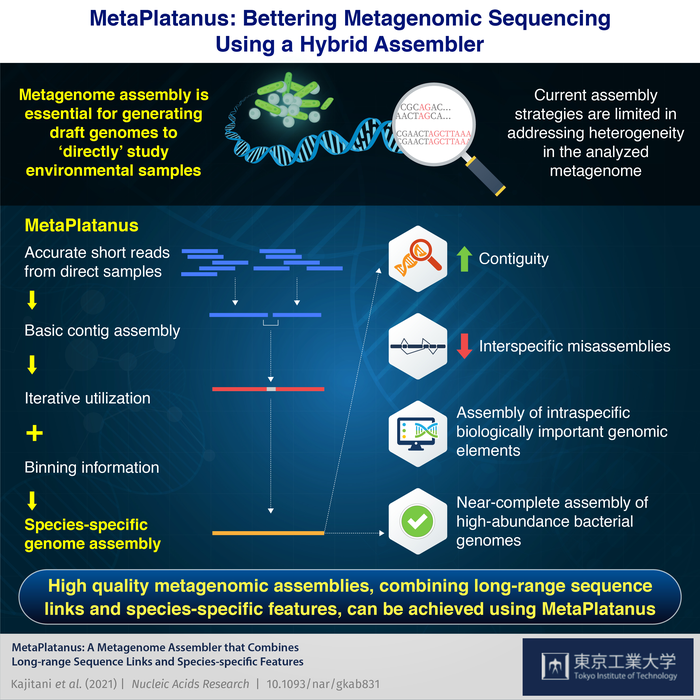
东京工业大学的科学家现在有了答案。他们开发了一种名为 MetaPlatanus 的新型宏基因组组装器,它可以生成准确的 DNA 序列,包括未培养生物的 DNA 序列。他们的突破性发现已作为研究文章发表在Nucleic Acids Research 上。
MetaPlatanus 使用准确的短 DNA 序列读取来组装重叠群。Contigs 是一段稍长的 DNA 序列,类似于较大基因组中的拼图。通过重复使用诸如长距离序列链接、物种特异性序列组成、覆盖深度和分箱信息等输入,重叠群被组装成更大的染色体尺度支架。
东京工业大学生命科学与技术学院助理教授、该研究的首席科学家 Rei Kajitani 博士解释了基于 MetaPlatanus 的支架生成的输入选择,他说:“我们采用混合宏基因组组装方法,不仅利用了短程和长程序列读取的优点,而且弥补了读取长度和样本本身带来的缺点。”
他进一步补充说:“我们已经将分箱应用于由难以组装的区域(例如重复区域)划分的链接序列。我们的方法是新颖的,因为到目前为止,分箱和组装过程的组合尚未作为公共工具实施!”
Kajitani 博士和他的团队不遗余力地检查 MetaPlatanus 生成的结果的准确性。在这方面,他们使用已知细菌的模拟数据集执行了一个称为基准测试的过程。毫不奇怪,MetaPlatanus 给出了高度连续的输出,很少有种间错误组装。
值得注意的是,在使用已发表的人类肠道数据测试 MetaPlatanus 的准确性时,它还组装了许多生物学上重要的元素,包括编码基因、基因簇、病毒序列和过半细菌基因组。
此外,与其他现有工具相比,只有 MetaPlatanus 能够对一些高丰度细菌基因组进行近乎完整的组装,同时使用已发布的人类唾液数据进行基准测试。
标签:
免责声明:本文由用户上传,与本网站立场无关。财经信息仅供读者参考,并不构成投资建议。投资者据此操作,风险自担。 如有侵权请联系删除!