大豆和玉米混合品系的同时遗传转化和基因组编辑
这项研究由拜耳作物科学研发部门植物生物技术和全球育种部门的跨职能团队开展。作者使用了一种名为混合品系转化和编辑(TREDMIL)的新方法,展示了大豆和玉米中多种优良基因型的同步转化和编辑。在这种方法中,在生产种子胚外植体之前将许多品系混合,然后使用基因组编辑机制转化混合品系,通过大豆 Dt1 特异性或玉米 Bm3 特异性 CRISPR RNA (crRNA) 和编辑核酸酶 Cas12a 的共表达,引导在目标基因区域产生插入/缺失。再生后,通过基因分型对品系身份进行解卷积,并通过扩增子测序验证编辑结果。
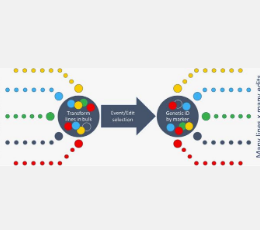
通过对再生事件进行基因分型,作者首先发现 104 个优良大豆基因型(101)中有 97% 同时发生转化,回收的转化子分布在从 00 到 VII 的不同成熟度组 (MG) 中。同样,40 个优良玉米雌性自交系(22)中有 55% 同时发生转化,转化子分布在从 92 到 117 的不同相对成熟度 (RM) 中。随后,作者使用扩增子测序证明,在代表所有 101 个转化品系的 94% 的大豆事件中检测到了Dt1靶位点的编辑,并且超过 80% 的转化大豆品系在该品系产生的 90% 以上的事件中产生了编辑。同样,在 69% 的玉米事件中发现了Bm3靶位点的编辑,代表了 22 个转化雌性自交系中的 17 个。这些结果清楚地证明了利用种子胚转化系统可以实现高效、基因型灵活的转化和基因组编辑。
作者进一步研究了 crRNA 靶位点Dt1和Bm3的 TREDMIL 实验的编辑结果,以了解不同种质的编辑情况。在三个Dt1靶位点上,1,506 个事件产生了 800 多个不同的编辑和 4,000 多个编辑。在三个Bm3靶位点上,96 个事件产生了 95 个不同的编辑和 200 多个编辑。种质中产生的大量编辑可以使基因组发现的速度比仅在单个种质中分析相同的编辑要快得多。此外,作者证明,在 98 种不同大豆基因型中,45%(1506 个中的 678 个)编辑事件中检测到了 Dt-1389 位点的明显 7 bp 缺失。类似地,在 65%(17 个中的 11 个)经过编辑的玉米雌性自交系中检测到 Bm3-2070 位点的明显 3 bp 缺失。在不同种质中创建相同的编辑可以提供一种机制,用于在育种计划中更早地评估种质与编辑之间的相互作用,这将使我们能够更快地对给定编辑的广谱功效做出决策。这些结果表明,TREDMIL 可以帮助加速未来精准育种中定制作物品种的开发和部署。
免责声明:本文由用户上传,与本网站立场无关。财经信息仅供读者参考,并不构成投资建议。投资者据此操作,风险自担。 如有侵权请联系删除!
-
【关羽骑赤兔包公铡太师打一生肖】在中国传统文化中,历史人物、典故与生肖文化常常交织在一起,形成许多有趣...浏览全文>>
-
【关羽连招技巧】在《王者荣耀》中,关羽是一位非常具有操作性的英雄,拥有高机动性和强大的控制能力。掌握他...浏览全文>>
-
【关羽历史上是怎么死的】关羽,字云长,是三国时期蜀汉的重要将领,以忠义、勇猛著称。他在《三国演义》中被...浏览全文>>
-
【关羽和刘备的马分别叫什么】在中国古代历史与文学作品中,三国时期的人物形象深入人心,尤其是关羽、刘备等...浏览全文>>
-
【关羽的媳妇】在历史与文学的交汇中,关羽作为三国时期的重要人物,其形象深入人心。然而,关于他的家庭生活...浏览全文>>
-
【关羽的青龙偃月刀现在在哪里】在中国古代历史和文学中,关羽是一位极具传奇色彩的人物,他所使用的“青龙偃...浏览全文>>
-
【关羽的青龙偃月刀多重】在中国古代文学和民间传说中,关羽是一位极具传奇色彩的英雄人物,他的形象不仅体现...浏览全文>>
-
【关于中秋节的英语手抄报】中秋节是中国传统节日之一,每年农历八月十五庆祝。它不仅是家人团聚的日子,也是...浏览全文>>
-
【关于中秋节的小故事】中秋节是中国传统节日之一,承载着丰富的文化内涵和深厚的情感寄托。自古以来,人们在...浏览全文>>
-
【关于中秋节的文化含义】中秋节,是中国传统节日中最具代表性的节日之一,通常在农历八月十五这一天庆祝。它...浏览全文>>
