新的理论模型验证了实验结果
一组从事理论和实验交叉工作的物理学家正在揭示分子马达的“团队合作”——称为 RNA 聚合酶 (RNAP)——介导 DNA 转录。在转录过程中,基因表达的第一步,RNAP 会“读取”DNA 序列并组装信使 RNA (mRNA),后者又作为生命必需蛋白质的模板。
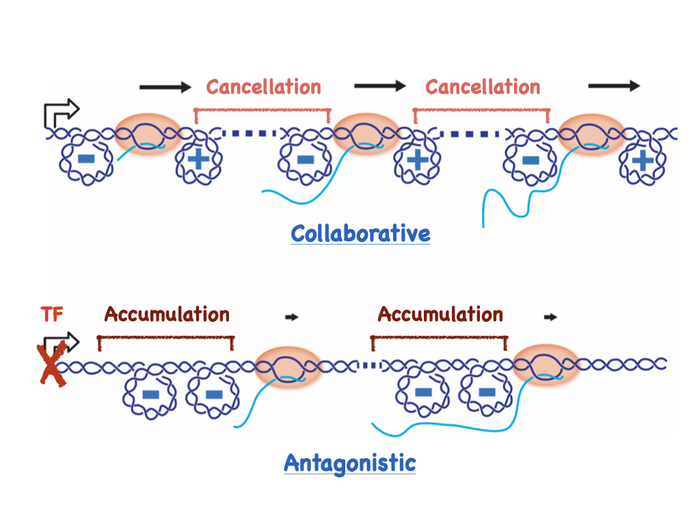
该团队包括主要作者 Purba Chatterjee,最近获得伊利诺伊州物理学博士学位。研究生,现为宾夕法尼亚大学博士后研究员;伊利诺伊州物理学名誉研究教授 Nigel Goldenfeld,现任加州大学圣地亚哥分校物理学杰出教授;和伊利诺伊州物理学教授 Sangjin Kim 介绍了一个新的理论模型,阐明了 DNA 中的超螺旋机制如何成为同时在 DNA 上进行转录易位的 RNAP 集体动力学的基础。RNAPs 动力学从合作模式切换到对抗模式,以响应细胞的需要。
这些发现于 2021 年 11 月 16 日发表在《物理评论快报》杂志上的文章“DNA 超螺旋驱动基因合成集体模式之间的过渡”中。
在转录过程中,当通过将螺旋的一部分解压缩成两条链而引入扭转应力时,DNA 超螺旋发生,其中一条将被转录。研究人员的工作首次揭示了模拟扭转下转录的两个基本要素:首先,众所周知的影响 RNAP 启动转录速率的转录因子也可以控制 DNA 超螺旋的传播,其次,存在的 RNAP 会影响单个 RNAP 所经历的扭转应力。
Goldenfeld 解释说,“超级螺旋对于任何曾经与花园软管或过去的电话线搏斗的人来说都是熟悉的。半刚性管,或者在这种情况下,螺旋难以折叠,它们弯曲成局部缠结 - 环“它看起来像八字形或更糟。生物学在活细胞内的 DNA 分子水平上与相同的几何问题作斗争。”
一旦 RNAP 启动转录,它就会沿链易位,组装 mRNA 的互补链。招募额外的 RNAP,每个 RNAP 沿着相同的 DNA 片段启动 mRNA 合成。随后的 RNAP 启动的速率通常由转录因子控制,转录因子是一种与 RNAP 启动转录位置的 DNA 位点结合的蛋白质。
先前的实验和理论研究预测,转录过程中 RNAPs 沿 DNA 易位的速度随着活跃转录相同序列的 RNAPs 的数量而增加,但在 2019 年,Kim等人。首次观察到,只要 RNAPs 以高于某个阈值的速率启动转录,无论总数如何,RNAP 易位的速度仍然很高。令人惊讶的是,他们发现一旦启动子关闭,即 RNAPs 停止启动转录时,RNAPs 的数量就会影响速度。在当前的工作中,该团队描述了超螺旋如何成为这些集体效应的基础。
免责声明:本文由用户上传,与本网站立场无关。财经信息仅供读者参考,并不构成投资建议。投资者据此操作,风险自担。 如有侵权请联系删除!
-
【广西南宁三甲医院列表有哪些】在广西南宁市,三甲医院作为医疗资源的重要组成部分,承担着区域内的疑难重症...浏览全文>>
-
【广西南宁建设学校】“广西南宁建设学校”是一所位于广西壮族自治区南宁市的教育机构,专注于建筑类及相关工...浏览全文>>
-
【广西哪里有机场】广西作为中国重要的旅游和交通枢纽省份,拥有多个机场,方便旅客出行。无论是前往南宁、桂...浏览全文>>
-
【广西名酒排名】广西作为中国西南地区的重要省份,不仅自然风光秀美,其酿酒文化也源远流长。虽然广西不像四...浏览全文>>
-
【广西民族大学专业】广西民族大学是一所具有鲜明民族特色和多学科协调发展的综合性大学,坐落于广西南宁市。...浏览全文>>
-
【广西米浆的制作方法】广西米浆是一种传统的米制品,常见于广西各地的早餐或小吃中,口感细腻、香滑可口,是...浏览全文>>
-
【广西美女最多的城市推荐】在广西这片美丽的土地上,不仅有壮丽的自然风光和丰富的民族文化,还孕育了许多气...浏览全文>>
-
【广西麻将怎么打】广西麻将是一种流行于广西地区的传统麻将玩法,其规则与常见的四川麻将、广东麻将等有所不...浏览全文>>
-
【广发银行和工商银行一样吗】广发银行和中国工商银行(简称“工行”)都是我国重要的商业银行,但它们在性质...浏览全文>>
-
【广发银行贷款条件】广发银行作为国内知名的商业银行之一,提供多种贷款产品,以满足不同客户的资金需求。无...浏览全文>>
