用几何指纹解开蛋白质表面相互作用的谜团
瑞士洛桑瑞士生物信息学研究所的研究人员使用了一种几何深度学习工具,该工具可生成蛋白质表面的“指纹”,以描述对蛋白质 - 蛋白质相互作用至关重要的几何和化学特征。在他们发表在《自然》杂志上的论文“蛋白质相互作用与学习表面指纹的从头设计”中,研究小组报告说,他们假设的“指纹”导致了分子识别和新型蛋白质相互作用的基本方面的捕获。总结团队结果的研究简报发表在同一期刊上。
蛋白质是生物学的物理机械,是允许有机生命发挥作用的齿轮和齿轮,弹簧和阀门。它是细胞如何工作,药物如何与生物系统相互作用以及大多数疾病与生物系统相互作用的地方。通过对这种机器的掌握,科学可以治愈大多数病症。
机器学习,以及蛋白质组学,基因组测序和分子生物学,在过去十年中极大地加速了蛋白质研究,以至于我们可以预测几乎所有现有的功能结构,在任何配置中设计新的结构,并合成制造任何可以想象的蛋白质,但有一个微小但重要的警告 - 结合位点。
蛋白质相互作用的方式依赖于基于表面化学和结构的两种特定的物理“锁和钥匙”相互作用。下面有一个外缘遗址和一个埋藏地点。埋藏部位完成蛋白质的工作,但要进入它,需要起始的外缘信号来打开结构以允许进入。
就蛋白质组学在过去十年中的发展而言,蛋白质的亲和力特征仍然难以捉摸。这部分是因为蛋白质是挑剔的和pH依赖性的,具有可变的边缘表面化学和结合位点,这些位点是条件和位点特异性的。
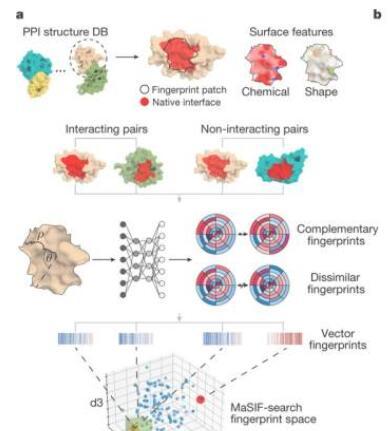
在目前的研究中,该团队通过基本上忽略除表面亲和力之外的所有内容来追求蛋白质之间的结构亲和力。撇开有关整体结构,功能和与其靶标相似蛋白质相互作用的信息,该团队将机器学习集中在蛋白质的表面相互作用以及确定两个分子相互作用最佳机会的几何和化学图案上,然后设计了适当的键。
通过计算蛋白质分子表面的指纹,该团队能够快速可靠地识别互补表面片段,这些片段可以在402.<>亿个候选表面内与特定靶标接合。
计算设计了几种从头蛋白结合剂,以参与四种蛋白质靶标:SARS-CoV-2刺突、PD-1、PD-L1和CTLA-4。一些设计经过实验优化,而其他设计则纯粹在数字空间中生成。结果是高度准确的亲和力预测,因为基于机器学习的粘合剂成功地参与了他们的目标。
作者指出,他们的框架可以“......除了提供在系统水平上研究生物过程中相互作用网络的手段外,还为药物设计、生物传感或生物材料等其他重要的生物技术领域开辟了可能性。
免责声明:本文由用户上传,与本网站立场无关。财经信息仅供读者参考,并不构成投资建议。投资者据此操作,风险自担。 如有侵权请联系删除!
-
【关于张起灵身世年龄介绍】在《盗墓笔记》系列小说中,张起灵是一个神秘而重要的角色,他的身世和年龄一直是...浏览全文>>
-
【关于张檬简介】张檬,中国内地女演员及歌手,1987年4月20日出生于山东省青岛市。她自出道以来,凭借扎实的演...浏览全文>>
-
【关于张謇的资料】张謇(1853年—1926年),字季直,号啬庵,江苏通州(今南通市)人,是中国近代著名的实业...浏览全文>>
-
【关于张家界百龙天梯的介绍】张家界百龙天梯位于湖南省张家界市武陵源风景名胜区,是世界上最高的户外电梯,...浏览全文>>
-
【关于张籍的资料】张籍(约766年-约830年),字文昌,是唐代著名诗人之一,与韩愈同为“古文运动”的重要人...浏览全文>>
-
【关于张海迪故事简介】张海迪,中国著名作家、残疾人代表,被誉为“中国的保尔·柯察金”。她自幼因病导致高...浏览全文>>
-
【关于张海迪的资料】张海迪是中国著名的残疾人作家、社会活动家,被誉为“中国保尔·柯察金”。她自幼因病导...浏览全文>>
-
【关于张飞的五个小故事】张飞是三国时期蜀汉的重要将领,与关羽并称“万人敌”,以勇猛、豪爽著称。在《三国...浏览全文>>
-
【关于张飞的故事】张飞是三国时期蜀汉的重要将领,与刘备、关羽并称“刘关张”,在《三国演义》中被描绘为勇...浏览全文>>
-
【关于鱼肉粥的做法】鱼肉粥是一道营养丰富、口感细腻的家常美食,尤其适合老人、小孩和病后恢复期的人群食用...浏览全文>>
