基因相互作用对肠道肿瘤进行分类以进行个性化医疗
根据今天发表在eLife上的研究,根据肿瘤细胞内常见的基因相互作用模式,肠癌可分为六种具有临床意义的亚型。
这种新的肠癌分类法将有助于了解这种疾病,并最终被医生用于精准医疗——帮助他们为个体患者量身定制最佳治疗方案。
“目前,疾病分期用于指导肠癌患者的临床管理,但这还不够,因为处于相似阶段的肿瘤之间可能存在很大差异,”主要作者,郑州大学第一附属医院硕士卢早渠解释说,郑州,中国。
“更详细的肠癌分子分类更有效,但它仍然基于某个时间点(例如,活检)肿瘤内基因活性水平的快照,并不能反映不断变化的动态基因活动以及基因如何相互作用。”
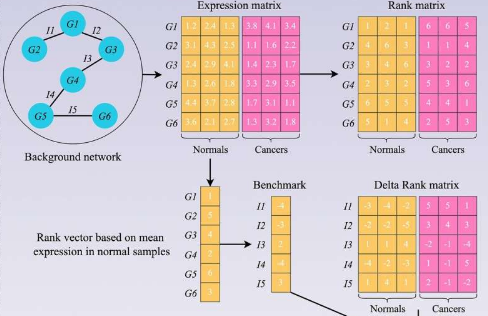
相比之下,生物相互作用网络——即基因通常相互作用的图谱——无论时间如何都保持相对稳定,并且可以更可靠地表征组织。此外,基因相互作用在正常组织中高度保守,但在疾病中发生了广泛的改变。通过量化不同基因对的“相互作用变化”,可以将患病组织中基因相互作用的总体变化与正常组织中背景水平的变化进行比较。
该团队应用这种基因相互作用网络 (GIN) 方法,使用 2,000 多个肠癌组织样本和 308 个正常肠组织样本来构建基因相互作用变化的大规模网络。然后使用该网络,他们能够将肠癌分为六种亚型,每种亚型都有不同的临床和分子特征。这些特征包括重要的肿瘤特征,例如癌细胞与其他细胞类型的比例,以及癌细胞是否与干细胞相似,这会影响肿瘤生长和扩散的可能性。
GIN 分类还确定了具有不同免疫原性的组——即肠肿瘤组织刺激免疫反应的可能性有多大,这对于确定患者是否可能对免疫治疗产生反应至关重要。其他有用的特征包括可能的预后、对放射疗法和免疫疗法治疗的抵抗力,以及对不同化疗药物敏感的可能性——所有这些都有助于调整治疗方案。当 GIN 方法在 19 个进一步的肠癌数据集中进行测试时,一致地确定了相同的六种亚型。
第一个 GIN 基于近 1,400 个基因之间的 2,000 多次相互作用,并产生了基于 289 个基因的分类器。这在临床环境中是不可能测试的,因此为了提供一种快速访问的临床工具,该团队将分类器简化为随机选择的 14 基因小型分类器。当他们对 214 个肠癌样本的子集使用迷你分类器时,他们发现它在识别六种肠癌亚型时与完整分类器一样稳定。
免责声明:本文由用户上传,与本网站立场无关。财经信息仅供读者参考,并不构成投资建议。投资者据此操作,风险自担。 如有侵权请联系删除!
-
【关于中秋节的英语手抄报】中秋节是中国传统节日之一,每年农历八月十五庆祝。它不仅是家人团聚的日子,也是...浏览全文>>
-
【关于中秋节的小故事】中秋节是中国传统节日之一,承载着丰富的文化内涵和深厚的情感寄托。自古以来,人们在...浏览全文>>
-
【关于中秋节的文化含义】中秋节,是中国传统节日中最具代表性的节日之一,通常在农历八月十五这一天庆祝。它...浏览全文>>
-
【关于中秋节的手抄报内容】中秋节是中国传统节日之一,象征着团圆与丰收。为了帮助同学们更好地了解中秋节的...浏览全文>>
-
【关于中秋节的诗句古诗】中秋节是中国传统节日之一,自古以来便有赏月、吃月饼、家人团聚的习俗。在众多文人...浏览全文>>
-
【关于中秋节的诗歌】中秋节是中国传统节日之一,自古以来就深受文人墨客的喜爱。许多诗人以中秋为题材,创作...浏览全文>>
-
【关于中秋节的诗词精选】中秋节,是中国传统节日中最具诗意的节日之一。自古以来,文人墨客常以中秋为题,写...浏览全文>>
-
【关于中秋节的来历100字】中秋节,又称月圆节,是中国传统节日之一,时间为农历八月十五。其起源与古代对月亮...浏览全文>>
-
【关于中秋节的句子】中秋节是中国传统节日之一,象征着团圆、思念与丰收。自古以来,文人墨客常用诗句表达对...浏览全文>>
-
【关于张起灵身世年龄介绍】在《盗墓笔记》系列小说中,张起灵是一个神秘而重要的角色,他的身世和年龄一直是...浏览全文>>
